Descodificando o xenoma

O xenoma é un código formado polas secuencias de catro nucleótidos representados polas letras G, A, C e T. Desde que se coñeceu, moita xente estivo investigando como descodificar o xenoma. E, por suposto, paira descodificar, primeiro hai que ler esas letras ou, dito doutro xeito, secuenciar o xenoma. É dicir, hai que saber en que orde están esas G, A, C, e esas T.
Paira iso utilizouse sobre todo un método nos últimos 30 anos, aínda que durante todos estes anos experimentou melloras significativas. Este método foi desenvolvido por Frederick Sanger e os seus compañeiros na década dos 70 --polo que recibiu o segundo Premio Nobel -.
A reacción básica do método de Sanger require catro compoñentes principais: a porción de ADN que se desexa secuenciar ou "patrón de ADN", catro tipos de nucleótidos libres, os pequenos filamentos de ADN de 20-30 nucleótidos coñecidos como iniciadores ou primeiros, e a encima de ADN polimerasa que sintetiza o ADN.
A reacción comeza quentando o patrón de ADN para que se separen os dous filamentos. Desta forma, o iniciador unirase a un dos filamentos (onde hai una secuencia adicional). Porque a polimerasa DNA alarga os filamentos de ADN, pero non se pode empezar pola nada, por iso é necesario o iniciador. Una vez que o iniciador uniuse ao patrón de ADN, o ADN polimerasa iniciadora comeza a estirarse nucleótido: onde hai una A en o patrón porá una T e viceversa, e onde hai una G una C e viceversa. Isto ocorre, ademais, sempre nun sentido determinado, é dicir, os dous extremos dun filamento de ADN denomínanse 5' e 3', e a polimerasa DNA engade nucleótidos ao extremo 3'.
Por iso, si partísemos de varias copias dun patrón de ADN, obteriamos outras tantas copias dun dos seus filamentos. Con todo, a clave do método de Sanger está no uso dalgúns nucleótidos modificados: os dideoxinucleótidos. A estes nucleótidos transformados fáltalles un grupo de hidroxilos no extremo 3', o que fai que non poidan engadirse outros nucleótidos.

Catro nucleótidos, catro reacciones
O método de Sanger require catro reacciones paira cada patrón de ADN. No catro reacciones atópanse o catro compoñentes mencionados anteriormente, pero en cada una delas colócase un único dideoxinucleótido. Así, por exemplo, una das reaccións conterá catro nucleótidos normais e algúns dideoxi-G. Nesta reacción, cando o ADN polimerasa iniciador comeza a estirarse, cada vez que necesita un G, poden ocorrer dúas cousas aleatorias: Tomar una G normal ou una dideoxi-G. A maioría son normais G, pero cando toma un dideoxi-G, a polimerasa non poderá seguir estirando a cadea. Así, a partir de varias copias do patrón, obteremos filamentos de diferente lonxitude, terminados todos nun G. E a partir de millóns de copias, finalmente teremos os filamentos correspondentes a cada G da secuencia. E o mesmo ocorre coas outras tres reacciones.
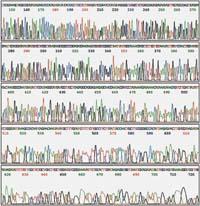
Posteriormente, os novos filamentos obtidos sepáranse por tamaños mediante unha electroforesis. Na electroforesis, os filamentos de ADN colócanse nun campo eléctrico e como o ADN ten carga negativa desprázanse do polo negativo ao positivo. No camiño colócase algo que dificulte este movemento, como un xel poroso, de maneira que as partes grandes tardan máis tempo en moverse dun polo a outro que as pequenas. Desta forma, pódense distinguir os filamentos que teñen un lado dun nucleótido.
Paira poder visualizar o resultado da electroforesis, os novos filamentos deben estar dalgunha maneira marcados. Inicialmente utilizábanse nucleótidos ou iniciadores marcados radiactivamente ou fluorescentes. Así, mediante unha autoradiografía, ou mediante raios ultravioleta, pódense ver como bandas, filamentos separados por tamaños.
Cada reacción consiste en trazar una liña na electroforesis, e o resultado final é que veremos una banda por cada nucleótido da secuencia nunha das catro liñas, e así, seguindo a orde de devanditas bandas, poderemos ler dita secuencia.

Automatización
Esta é basicamente a técnica utilizada paira secuenciar a maior parte do ADN secuenciado até o momento. Pero desde a súa fundación, Sanger experimentou importantes melloras. Un dos máis importantes foi a secuenciadora automática de ADN inventada por Leroy Hood en 1986. Hood marcou cada un do catro dideoxinucleótidos de forma que proporcionase una fluorescencia de diferente lonxitude de onda (cor). Así, por unha banda, en lugar de catro reacciones, basta cunha soa, e por outro, a lonxitude de onda emitida ao ser iluminada con raios ultravioleta pódese detectar automaticamente.
Nos secuenciadores automáticos, cada mostra vai nunha soa liña, detectándose o catro nucleótidos pola súa diferente cor. Nos secuenciadores modernos, os filamentos de ADN sepáranse pasando por uns capilares de fibra de vidro, á saída do extremo son golpeados por un láser e a máquina detecta a presenza de fluoerescencia. Os filamentos que salguen primeiro serán o iniciador máis un nucleótido e a continuación sairá toda a secuencia.
Durante a última década, os secuenciadores automáticos de ADN melloraron considerablemente, podendo procesar moitas máis mostras con maior rapidez e facilitando o seu uso. Na actualidade pódense secuenciar centos de mostras nunha sesión e nun día pódense realizar unhas 24 sesións.
Xenoma grande

Con todo, aínda non é tarefa fácil descodificar todo un xenoma. As técnicas de secuenciación do ADN serven paira secuenciar pequenos fragmentos de ADN, secuencias de 300-900 nucleótidos. De feito, en filamentos máis longos, non poden separarse con precisión da cara dun nucleótido.
Pero o xenoma pequeno dunha bacteria tamén contén millóns de nucleótidos, e o humano 3.000.000.000, dividido en 23 cromosomas. Imaxínache que paira escribir todo o xenoma letra a letra nesta revista necesitariamos unhas 20.000 revistas. A lonxitude media das secuencias lidas (500 nucleótidos) sería o seguinte parágrafo, e en 20.000 revistas inclúense seis millóns de parágrafos deste tipo. Ademais, paira conseguir una fiabilidade suficiente, una vez non, habería que ler entre 6 e 10 veces todas estas revistas.
É o que fixeron no Proxecto de Xenoma Humano. Por partes leron todo o xenoma humano letra a letra. Con todo, dividir todo o xenoma e ler todas estas partes é una cousa e a outra é saber a orde no que se deben unir esas partes.
O proxecto comezou coa construción dun mapa do xenoma. Paira poder percorrer os cromosomas sen perder, tomáronse miles de puntos de referencia. Una vez realizados estes mapas, creouse una "biblioteca" de fragmentos de ADN que englobaría todo o xenoma. Paira iso, estes anacos de ADN foron almacenados no interior das bacterias, e grazas aos puntos de referencia, cada parte do xenoma era coñecida. En definitiva, é o que ofrece calquera biblioteca: información ordenada. Isto permitiu traballar de forma coordinada en laboratorios de todo o mundo.
Os fragmentos de ADN foron almacenados nas bacterias E. coli que viven habitualmente nos nosos intestinos, como cromosomas bacterianos artificiais (BAC). En cada BAC almacénanse fragmentos de ADN de 100.000 ou 200.000 nucleótidos.
As bacterias E. coli conservan o tempo que queiras no conxelador. Así, cando o científico necesita un BAC da biblioteca, a bacteria só ten que resucitar a 37ºC. Ademais, cando a bacteria que contén o fragmento de ADN reprodúcese con el. Así, abonda con que as bacterias deixen crecer una noite paira obter millóns de copias da súa parte interna de ADN. A isto chámaselle amplificación do ADN.
Os BACs son aínda moi grandes paira poder secuenciar. Por iso, os BACs tamén se dividen aleatoriamente, obtendo partes máis pequenas que se solapan. Estas partes introdúcense nos virus ou plásmidos que infectan as bacterias paira o seu posterior amplificación nas bacterias E. coli. Por último, o ADN das bacterias é purificado e secuenciado. A continuación identifícanse as secuencias que se solapan nas distintas partes e complétase a secuencia completa do BAC. Ao mesmo tempo, os BACs tamén se superponen. E así, a lectura por lectura, o BAC por BAC e o cromosoma por cromosoma, até ler todo o xenoma.
Carreira xenómica
Ler así un xenoma completo require moito tempo, traballo e diñeiro. Por iso, os investigadores están a investigar continuamente paira desenvolver técnicas de secuenciación máis rápidas e baratas. Moitos novos métodos buscan aumentar a capacidade de secuenciación e realizar miles ou millóns de secuencias simultaneamente. Pero tampouco faltan ideas novas, como un método que detecta no momento en que a dna-polimerasa engade o nucleótido ao filamento, ou que detecta correntes eléctricas que identificarían cada nucleótido ao pasar os filamentos por nanopors.
Os proxectos son moitos e estanse desenvolvendo a unha velocidade vertixinosa. Non é de estrañar que as institucións públicas e privadas estean a investir moito. En 2006, o National Human Genome Research Institute (NHGRI) de EE.UU. achegou 13 millóns de dólares ao financiamento de proxectos paira acelerar o desenvolvemento de tecnoloxías que abaraten a secuenciación de ADN. Ese mesmo ano, a Fundación X Prize anunciou un premio de dez millóns de dólares paira o primeiro equipo que en 2006 realiza un dispositivo capaz de secuenciar cen xenomas humanos en dez días. E, cen non, un proxecto internacional que acaba de lanzar pretende secuenciar os xenomas de mil seres humanos.
Díxose que a secuenciación do xenoma é un dos avances máis importantes na historia do ser humano. Investigadores de todo o mundo tentan desvelar todos os segredos do xenoma e descodificar o código. Está claro que estamos na era da xenómica.






