Peces, genes y chips: libertad de expresión ante la contaminación

(Foto: Laboratorio de Pez Cebra de ZFIN y Oregón)
A medida que aumenta la contaminación, cada vez es mayor la necesidad de conocer cuáles son los daños que estos contaminantes provocan en los organismos, cómo condicionan el comportamiento del organismo y cómo pueden preverse estos efectos. Para el estudio de estos daños, aunque los estudios se han centrado en varios niveles, los realizados a nivel molecular se han materializado, promovido y hecho imprescindible al conocer toda la secuencia genómica de los seres eucariotas (mosca de fruta, lombriz de tierra o ser humano).
La razón de esta importancia radica en la función y características del propio genoma, que puede ser considerado como una biblioteca que alberga toda la información (fenotipo) para formar cada organismo. Esta biblioteca está organizada en libros o genes que almacenan información para producir proteínas que cumplen la mayoría de las funciones de la célula. Así, los cambios en los genes y/o en la expresión de estos genes pueden provocar alteraciones en el funcionamiento normal de la célula.
Regulación de la expresión génica
Las diferencias fenotípicas entre especies (arquitectura corporal, metabolismo, comportamiento...) deben entenderse en un doble nivel. Por un lado, cada especie tiene su propia biblioteca (genoma); algunas especies están dotadas de numerosos libros (genes) y otras menos. Por otro lado, y como acabamos de conocer, los genomas del hombre y del mono están dotados de forma similar, por lo que otra cosa debe condicionar las diferencias notables entre estas dos especies. En esta segunda variable participa la regulación de la expresión génica.
En gran medida, los libros que tenemos en la biblioteca los leemos de una u otra manera. Eso queda claro si miramos nuestro colco. Los seres humanos somos seres pluricelulares y, aunque todas nuestras células tienen el mismo genoma, es difícil marcar las semejanzas entre un hepatocito y una neurona, ya que las células durante el desarrollo, según las condiciones del medio, expresan (leen) determinados genes (libros). Por ello, para ganar en la competición entre especies utilizamos las vías metabólicas de cada momento, siempre con el objetivo de obtener el máximo rendimiento energético.
Por ejemplo, los habitantes del primer mundo hacemos tres comidas al día. Por tanto, para cumplir con nuestras obligaciones energéticas, por un lado recurrimos al metabolismo de los hidratos de carbono a través de las enzimas de la glicolisis, pero por otro lado acumulamos lípidos a través de la producción de triglicéridos. Por ello, no necesitamos el catabolismo de los lípidos hasta que sentimos hambre. Cuando se inicia una huelga de hambre, por ejemplo, los genes productores de enzimas procedentes de las vías metabólicas que intervienen en el catabolismo de los triglicéridos comienzan a expresarse rápidamente. Es lo que se conoce como regulación de la expresión génica.


Para llevar a cabo esta regulación, los seres pluricelulares están dotados de proteínas especiales, factores de transcripción, que trabajan en la exploración continua del medio celular. La función de estas proteínas es hacer frente a los cambios que se producen en el medio (cambios en la concentración de los alimentos, factores fisiológicos, hormonas, parásitos, enfermedades...) mediante cambios en la expresión génica.
Todos los compuestos contaminantes y tóxicos presentes en el medio son, por tanto, una variable ambiental para estos factores de transcripción. Pondremos un ejemplo: en la mayoría de los animales se pueden distinguir ejemplares masculinos y hembras. Las principales diferencias entre machos y hembras dependen de dos hormonas en la mayoría de las especies: la testosterona en los machos y el estradiol en las hembras. El estradiol pone en marcha alternativas de desarrollo representativas en las hembras mediante un factor de transcripción denominado receptor de estrógenos. Esto provoca la producción de vitelogenina, la lipoproteína de reserva más importante de los ovocitos de las peces hembras (también en otros grupos). Producen peces hembras, no machos, aunque también tienen el gen que produce vitelogenina.
En grandes cantidades llegan a nuestros ríos compuestos con una estructura química similar al estradiol, como los alquilfenoles de jabones utilizados en nuestras limpiezas, los ftalatos utilizados en plásticos o las hormonas artificiales que tomamos en las pastillas anticonceptivas.
Estos compuestos transcriben a través del receptor de estrógenos el gen que codifica la vitelogenina en los machos. Pero el gen de la vitelogenina no es el único que responde; los compuestos estrogénicos dados a un macho influyen en la expresión de cientos de genes. En consecuencia, existen en el mundo fluvial, donde los peces machos producen proteínas marcadoras de las hembras, en las que junto con los espermatozoides también se han encontrado ovocitos en algunos casos. Este ejemplo pone de manifiesto, por tanto, los efectos significativos que sobre las poblaciones de muchos organismos pueden tener algunos compuestos de uso cotidiano.
Toxicología y genómica juntos
Uno de los objetivos actuales sería, por tanto, conocer las obligaciones de todos estos genes de diferentes genomas en la célula. Aunque todavía estamos bastante lejos, año tras año conocemos el genoma de más organismos y conocemos cada vez más claramente cuáles son los mecanismos de regulación de la expresión génica. Por tanto, la toxicogenómica tiene como objetivo investigar qué mecanismos de regulación de la expresión génica ponen en marcha ciertos contaminantes.

Se sabe que no todos los individuos expuestos a contaminantes ambientales responden de la misma manera. Esta respuesta está limitada por factores internos del propio huésped (estado de desarrollo, carácter genético, hormonal, metabólico...) y ambientales (concentración del contaminante y tiempos de exposición). Por tanto, en función de la combinación entre contaminante, presencia de cofactores y sensibilidad genética, dos individuos o dos células del mismo individuo pueden dar respuesta diferente a un efecto determinado.
La respuesta de los organismos ante una determinada sustancia tóxica es consecuencia de las interacciones de la expresión de diferentes genes. Por ello, en los últimos años cada vez son más los laboratorios que están estudiando estos genes. De ahí nació la toxicogenómica.
En toxicogenómica, el análisis de la expresión génica, además de informar sobre el estado de las células, nos muestra cómo responden estas células ante diferentes estímulos. Por lo tanto, en primer lugar, deberíamos conocer la secuencia genómica del organismo de interés o, al menos, los agobios en los que las variables del medio pueden expresarse en exceso o en exceso y en qué vías metabólicas intervienen.
En los últimos años se ha avanzado enormemente en este campo y es conocido en gran medida por los diferentes tipos de compuestos, como los que provocan la inflamación, los compuestos estrogénicos, los inmunosupresores o los carcinogénicos, que modifican la expresión de los genes. Así, por ejemplo, cuando hablamos de estrogenicidad, sabemos que los principales genes a analizar son los que codifican la vitelogenina (en los peces) y las proteínas de la matriz extracelular del huevo; cuando hablamos de inmunosupresores, la interleucina, la proteína del complemento o varios tipos de citoquinas; y cuando hablamos de carcinogénicos, junto con los genios Bcl-2, p53, c-myc, etc.
A la hora de determinar cuáles son los genes a estudiar, el trabajo de los futuros toxicológicos sería investigar los cambios expresivos de estos genes frente a los contaminantes. Sin embargo, en casos desconocidos, los toxicólogos deberían buscar nuevos genes que respondan a los efectos del contaminante.
Uso de peces en toxicogenómica ambiental

Decir que los peces son los mejores organismos a la hora de analizar la toxicología ambiental no es decir demasiado, ya que estos vertebrados tienen el cuerpo sumergido en el agua y, por tanto, están directamente relacionados con los contaminantes presentes en el medio, ya sea a través de las branquias o del sistema intestinal hepático. El grupo de peces es un grupo amplio, tanto en el número de especies como en el número de individuos dentro de la especie, lo que la convierte en un grupo de organismos con diferentes fisiologías, formas de vida y diversos sistemas de adaptación.
Esta diversidad también se refleja en el genoma, ya que los peces presentan un genoma mucho más plástico y cambiante que otros vertebrados. Por tanto, ante los cambios que se producen en el medio, la contaminación, especialmente el pescado, es más sensible que otros muchos organismos. Asimismo, la utilización del pescado como modelo nos llevará a comprender mejor el genoma de los mamíferos a la hora de interpretar los genomas de otros taxones. Además, no hay que olvidar que muchas especies de peces, además de su importancia ecológica o científica, tienen también una importancia económica, especialmente en aquellos países en los que el pescado es un alimento habitual.
Así, actualmente se trabaja con numerosas especies de peces, entre ellas el pez cebra, la medaca, la carpa, la trucha, el salmón y la platija. Aunque nuestro conocimiento del genoma de unos y otros es radicalmente distinto, en la actualidad (noviembre de 2005) conocemos más de 10.000 secuencias de genes que se expresan en 14 especies de peces teleósteo. Además, la secuencia completa del genoma de los teleosteos Fgu rubripes , Danio rerio y Tetraodon nigroviridis ya es conocida.
Microchips: herramientas para investigar la expresión génica
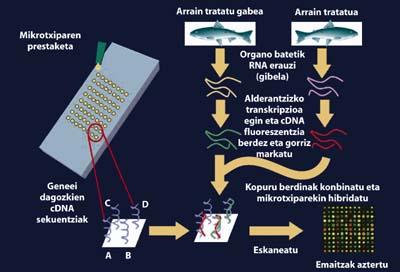
Tenemos peces y genes que queremos estudiar, ¿qué hacer con ellos? ¿Cuál es la metodología a seguir? Los microbildumas de ADN o microchips son herramientas de trabajo actuales y futuras basadas en el análisis holístico de la expresión génica, cada vez más conocidas. Estos chips están formados por cientos o miles de sondas de ADN correspondientes a genes de interés unidos en un soporte sólido.
Los microchips tienen sólo el tamaño de las portas de vidrio que se utilizan en el microscopio y nos permiten detectar simultáneamente todas las secuencias que nos interesan y son conocidas. Con ellos se pueden detectar moléculas adicionales de ADN y ARN marcadas con diferentes marcadores. Para ello, se extrae el ARN de cada uno de los órganos de los peces (como el hígado) tratados con contaminantes y no tratados, y se sintetiza, mediante transcripción inversa, el ADN auxiliar (cDNA). Este cADN constituye la transcriptoma de la célula, es decir, la recopilación de la expresión génica del genoma de ese animal en ese momento. Este cDNA se puede marcar con marcadores fluorescentes, uno en verde y otro en rojo, en definitiva, con el objetivo de diferenciar el material genético de ambos organismos. A continuación se combinan las mismas cantidades de cDNA de los organismos que queremos analizar y comparar y se hibrida la mezcla con microchip. Así, si en el momento de la extracción en el pescado tratado se estaba expresando mucha vitelogenina y en el momento de la extracción en el que no se trataba, se podría detectar esta diferencia por los diferentes colores que el chip va a presentar para ese gen.
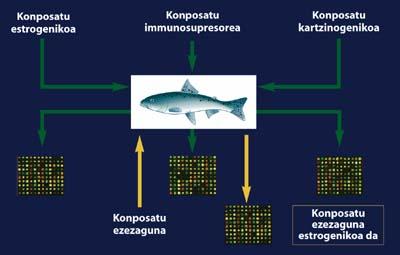
En los experimentos de laboratorio que se han llevado a cabo a lo largo del mundo, determinados tipos de peces se han mantenido expuestos a ciertos compuestos tóxicos. En estos casos se han desarrollado microchips sobre alguno de los órganos del pescado, por lo que ya se conoce el gen que responde a la exposición de estas especies a este tipo de contaminantes.
Estos patrones de expresión no difieren mucho de los conocidos en los mamíferos. En la actualidad, si alguno de estos organismos que conocemos cómo cambia el patrón de expresión de los genes se encuentra bajo la influencia de un compuesto desconocido en el medio, tendríamos la capacidad de identificar el tipo de contaminante respecto a los patrones de expresión que conocemos. De esta forma, antes de que este contaminante pueda causar daños a los seres vivos, tendríamos la oportunidad de tomar medidas locales para evitarlos. De la misma manera, una vez fabricadas nuevas drogas y compuestos, podemos prever los cambios que estos puedan producir a los organismos antes de su puesta en el mercado.
Mecanismos moleculares de contaminantes y análisis de la calidad ambiental
En los últimos años los laboratorios que aplican chips a los peces son cada vez más numerosos, tanto en experimentos de laboratorio como en poblaciones rurales. En los estudios que se están realizando en el Reino Unido, por ejemplo, se está tratando de determinar nuevos biomarcadores moleculares que respondan a contaminantes específicos y definir cuáles son sus mecanismos.
Así, se han comparado las expresiones génicas de los peces platija que habitan en la árida ría llamada Alde y en la contaminada ría de Tyne. Para ello, tras clonar en la platija genes relacionados con diferentes tipos de estrés en otras especies, han diseñado un microchip de 13.000 secuencias. Tras hibridar el cDNA de los peces de ambas zonas con chipis, se han encontrado diferencias expresivas significativas en cien secuencias. Los más representativos de los que aparecen inducidos en Tyn se han relacionado con la presencia de altas concentraciones de hidrocarburos policíclicos aromáticos en el medio CYP1A1, paraoxonasa, (enzimas de la fase I del metabolismo de detoxificación), UDPGT y aldehído deshidrogenasa (enzimas de la fase II). Los cambios encontrados en el resto de genes, sin embargo, responden al estrés global.
Además, han observado que en el estradiol se inducen, entre otros, los genes que codifican la vitelogenina y la coriogenina. Algunos de estos cambios expresivos se han descrito en otros experimentos de laboratorio recientes. Por ejemplo, en los machos de platija mantenidos durante 21 días bajo el estradiol de etinil, más de 200 genes manifestaron diferente expresión en animales tratados. Cabe destacar que entre estos genes se encuentra inducida la expresión del gen que codifica la vitelogenina, la UDPGT y la proteína de la matriz extracelular de ovocitos, ZTP.
Por tanto, los microchips de ADN son potentes herramientas para caracterizar las propiedades toxicológicas de las sustancias químicas. Como cualquier otra técnica en crecimiento, todavía son necesarias numerosas investigaciones para poder definir y considerar el uso de microchips de ADN en el campo/entorno desde el punto de vista de la ecotoxicología. Sin embargo, los microchips son fotografías exactas de la expresión génica de los organismos y pueden mostrar claramente cómo la fisiología del pescado se reorganiza bajo los efectos de los cambios en el medio del pescado.
BIBLIOGRAFÍA




