L'ADN ancien, nouveau champ de recherche
Lors de cette rencontre de deux jours, les découvertes de chaque groupe ont été dévoilées et l'avenir de la recherche de l'ancien ADN a été analysé. Parmi les participants se trouvait la professeure et chercheuse du campus de Leioa de l'UPV, Conchi de la Rúa, avec qui nous avons pu entretenir une conversation. Il nous a expliqué les recherches qui ont été menées dans un monde unique et son expérience.
Qu'est-ce que l'ADN ancien ?

La majeure partie du matériel génétique d'un organisme est stockée dans le noyau de la cellule comme chromosome. Les chromosomes sont constitués de 3.000 millions de nucléotides qui codent entre 50.000 et 100.000 gènes. « Cependant, la majeure partie de la chaîne ADN n’est pas un encodeur de gènes et ce caractère neutre fait que l’ADN sert à la recherche génétique », explique Conchi.
Si nous comparons l'ADN ancien à l'ADN des êtres vivants, il y a quelques différences notables. Tout d'abord, la quantité d'ADN ancien obtenue dans les extractions est très faible par rapport à celle que l'on peut obtenir d'un vivant, entre 0,1 et 1%. Deuxièmement, la chaîne ADN résultante est fragmentée en chaînes de 100 paires de bases, tandis que chez les êtres vivants, on peut obtenir des chaînes de milliers de paires de bases.
Ces différences sont dues aux processus de dégradation qui se produisent dans les échantillons. « Les effets d’oxydation et d’hydrolyse qui se produisent après la mort de l’organisme provoquent la rupture de la chaîne d’ADN, la conservation de l’organisme étant d’une grande importance pour la qualité des échantillons dans un environnement sec et sans oxygène », explique-t-il. Pour cette raison, l'âge limite de l'ADN ancien est de 100.000 ans.
ADN mitochondrial
En raison du nombre limité d'anciennes molécules d'ADN récupérées, la plupart des chercheurs se sont adressés à travailler avec l'ADN mitochondrial dans le cas de l'ancien ADN. Comme l'ADN nucléaire est unique dans chaque cellule, l'ADN mitochondrial est abondant. Cette abondance augmente la survie et les chances de récupération de l'ADN.
Dans le cas de la mitochondrie, la longueur de la chaîne ADN est de 16.500 paires de bases azotées, ce qui représente 0,0005% du génome humain. L'ADN des mitochondries humaines et de plusieurs vertébrés est séquencé et dans cette séquence se trouve une zone appelée zone de contrôle. Cette zone équivalente à la base 1.100 est neutre, de sorte que les mutations apparaissent uniquement sous l'influence du temps et non par la sélection naturelle. En outre, le point de contrôle supporte 90% des mutations d'ADN mitochondrial.

A ces particularités il faut ajouter une autre: L'ADN mitochondrial est seulement reçu de sa mère. « L’ADN mitochondrial se trouve dans la queue des spermatozoïdes, et pendant la fécondation de l’ovule, la queue est à l’extérieur, dit Conchi, laissant ainsi l’ADN mitochondrial masculin ».
Cela signifie réduire de moitié la zone d'étude et faciliter le suivi de la marque de dérive génétique.
Initiation
La première recherche de l'ADN antique a été réalisée en 1984 avec un animal qui a disparu il y a 150 ans. Une ancienne séquence d'ADN a été obtenue pour la première fois du tissu musculaire sec d'un equus quagga. De cette séquence, ils ont démontré la relation évolutive entre le quagga et les parents les plus proches d'aujourd'hui, le zèbre.
Un an plus tard, en 1985, d'autres chercheurs ont récupéré l'ADN des cellules superficielles des momies. Sur les 110 momies sélectionnées, 23 étaient mieux conservées et seulement deux d'entre elles ont obtenu l'ADN. Cette étape a été le début de l'étude de l'ancien ADN humain.
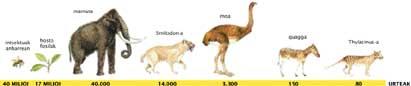
En poursuivant les recherches, au milieu des années 90 on réussit à séquencer des fragments d’ADN de vertébrés déjà disparus, comme le Thylacinus ou le loup martsupial (120 ans), le Smilodon ou le tigre «aux dents de sabre» (14.000 ans), le mammouth foncé (10.000-50.000 ans) et le model (3.300 ans).
En outre, une feuille de magnolia appelée Ma (17 millions d'années) et plusieurs insectes stockés dans l'ambre ont été étudiés, mais leurs études ne sont pas actuellement considérées comme fiables. « Il n’a pas été possible de répéter les résultats obtenus dans ces laboratoires ni dans le laboratoire, ni dans d’autres, ce qui invalide les résultats », affirme Conchi. «Cependant, il a été démontré que l’ADN ancien était récupérable.»
En 1986, une étape plus importante est franchie en lançant la nouvelle méthode d'analyse PCR. Cette technique a révolutionné la recherche de l'ADN et a permis la découverte des gènes mitochondriaux.
Méthode PCR
La réaction en chaîne ou méthode PCR de la polymérase consiste à synthétiser des millions de copies d'un petit fragment d'ADN. Dans ce processus cyclique
Une partie de la chaîne ADN est multipliée de façon exponentielle jusqu'à ce que des échantillons suffisent pour appliquer les différentes méthodes d'analyse. Le PCR nécessite la conception de courtes chaînes ADN avec fonction limitante, complémentaires de la chaîne ADN objet d'étude. La fonction de ces limiteurs est de fixer la longueur de la chaîne ADN à dupliquer, car ils sont ajoutés au début et à la fin. Ensuite, l'enzyme appelée Taq polymérase effectue le doublage de la chaîne ADN.
Ainsi, la méthode PCR a permis d'étendre les études aux os et aux dents et, étant un matériau courant provenant de fouilles archéologiques, la porte a été ouverte à l'étude de l'histoire biologique des siècles.
Problèmes méthodologiques et pollution
Cependant, la capacité de duplication de la méthode PCR devient un problème dans le cas de l'ancien ADN. L'ADN ancien a un risque élevé de contagion en entrant en contact avec n'importe quel élément externe. Compte tenu de cela, et puisque les anciennes chaînes d'ADN qui sont récupérées sont courtes, la méthode PCR a une grande probabilité de copier des fragments d'ADN externes. Il est donc impératif de prendre une série de mesures pour assurer le résultat du doublage.
Tout d'abord, l'isolement physique du laboratoire doit être assuré afin qu'il n'affecte pas les éléments extérieurs; deuxièmement, il faut utiliser uniquement l'équipement destiné à la récupération de l'ADN ancien et, finalement, il faut assurer la stérilité totale des échantillons à utiliser. Ces échantillons sont généralement extraits des grains qui sont le mieux conservés, généralement en utilisant des os et des dents. “La quantité d'ADN ancien disponible extraite de la pulpe est plus grande que celle obtenue à partir des autres tissus ou os, et aussi dans les gisements est généralement une pièce commune”.
En plus de toutes les mesures mentionnées, les études doivent être répétées dans le même laboratoire et dans différents laboratoires, en utilisant plus d'un échantillon du même exemplaire, ce qui assure l'existence ou non de contamination. Enfin, pour homologuer l'ancienne séquence d'ADN, il doit être comparé à l'univers de séquences connues qui sont stockées dans les bases de données, analysant ensuite les résultats dans un contexte phylogénétique approprié et les comparant aux séquences d'autres espèces vivantes ou mortes d'un même groupe taxonomique.
Quelles méthodes utilisez-vous pour les examens?
« Deux méthodes, une méthode de séquençage et une méthode d’analyse des enzymes restrictives peuvent être utilisées. »

Le séquençage consiste à connaître la localisation des bases azotées responsables de l'information génétique dans la chaîne ADN. Cette méthode automatique à plusieurs étapes consiste en une amplification spéciale de la chaîne ADN, où des fragments fluorescents de différentes tailles sont obtenus. Ils sont ensuite distribués par électrophorèse, c'est-à-dire séparés les uns des autres dans un gel sur lequel un champ électrique est appliqué. La vitesse de migration dépend de la taille. Dans ce processus, un lecteur laser détecte l'émission fluorescente de chaque partie, qui dépend de la dernière base azotée qui a été ajouté dans le processus de PCR. Par conséquent, la base azotée est connue en fonction de la fluorescence dégagée.
Avec les enzymes restrictives, on analyse les séquences cibles. « Les séquences d'arrivée sont de courtes chaînes de couple azotées dans la chaîne ADN. » Les enzymes restrictives sont capables de briser la chaîne ADN d'où se trouvent les séquences cibles. Ces séquences d'objectif, qui ont une longueur de 4-6 paires de bases azotées (pb), sont différentes pour chaque enzyme réductrice, c'est-à-dire qu'une enzyme réductrice doit trouver une séquence d'objectifs pour pouvoir briser la chaîne d'ADN, qui n'existe souvent pas. « En voyant si la chaîne ADN réagit ou non avec différentes enzymes, nous pouvons savoir si elle a différentes séquences cibles et s’il s’agit ou non de séquences cibles, selon les différents modèles existants, nous pouvons voir des génétiques relationnelles ». Méthodologiquement, cette deuxième technique est plus simple et plus rapide, ce qui permet d'obtenir plus de résultats positifs.
Futur proche
Ces dernières années, la recherche de l'ADN ancien a porté sur la recherche de relations phylogénétiques, l'étude de la variabilité génétique au fil du temps ou la recherche de changements démographiques. Tout cela est dû aux avancées en biologie moléculaire et au développement de la technique PCR. Cependant, il reste encore des obstacles tels que le risque de contamination et la mauvaise conservation de l'ADN ancien.
Pour l'avenir, l'étude vise à améliorer la méthodologie d'extraction de l'ADN et à augmenter le nombre d'anciens échantillons. De plus, les nouvelles recherches se concentreront sur l'approfondissement de la recherche sur l'ADN nucléaire. Ces indicateurs permettront non seulement d'analyser l'information reçue de sa mère, mais aussi celle de son père. L'avenir nous donnera-t-il la limite de cette science ?
I Espagne de l'ADN antique. Congrès Congrès Congrès Congrès Congrès CongrèsLa Résidence de la Vitrera de l'Université Autonome de Madrid, située à Miraflores de la Sierra, a accueilli la première édition espagnole de l'ADN ancien. Congrès. Parmi les invités se trouvaient des experts de 15 centres de recherche dédiés à la biologie évolutive, paléobiologie, anthropologie, génétique moléculaire, médico-légale et ictologie. Ces groupes de chercheurs ont envisagé de connaître et d'approfondir les objectifs communs de leur science. Parmi ces objectifs, on a analysé la création d'un groupe de travail sur l'ADN ancien. Ce groupe se chargerait d'établir une procédure de base pour garantir la qualité des recherches dans cette science. Un autre objectif était de renforcer les relations entre les laboratoires, en encourageant la collaboration entre les groupes chercheurs. Enfin, il a commenté l'intention de faire connaître les applications potentielles de ce domaine scientifique afin que les gens prennent en compte les applications possibles de la recherche de l'ADN ancien dans d'autres domaines. |
Qu'y a-t-il en Euskal Herria ?
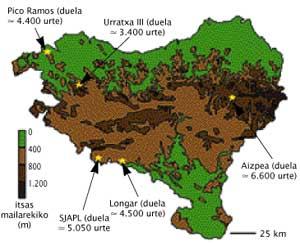
Conchi de la Rúa et son équipe dirigent leurs recherches vers l'anthropologie. La méthode des enzymes restrictives a permis de classer 97% des échantillons exempts de contamination de divers gisements basques. Bien que le pourcentage semble élevé, il faut dire que seulement 25% des échantillons récupérés sont représentés, car seuls les dents ont été analysées en parfait état pour éviter le risque de contamination.
Parmi les découvertes ont été obtenus des données pour s'opposer aux théories de certains chercheurs italiens. Ces chercheurs italiens rapportaient un groupe qui, il y a entre 10.000 et 15.000 ans, s'est étendu du Pays Basque à l'Europe. Le but de cette théorie était d'expliquer l'abondance d'un groupe génétique existant actuellement dans la société basque, le soi-disant V Haplotalde. Les travaux des chercheurs basques ont montré qu'il y a l'absence de ce groupe dans les anciens échantillons d'ADN, c'est-à-dire qu'il n'y a pas d'indications de cette époque du haplotalde. Que signifie cela ? “Que ce groupe de Haplots n'a pas été créé ici parce que si c'était ici, il devrait apparaître sur des échantillons de moins de 10.000 ans”.
Conchi et ses compagnons mettent le point de vue sur la dérive génétique pour expliquer l'abondance de ce groupe. « La mutation qui définit le Haplotaldea a pu atteindre un taux élevé dans un groupe de population, comme c’est le cas de Gipuzkoa, à une époque où la population était réduite, et les croissances de population plus tard ont permis d’étendre cette mutation ».





