Descodificant el genoma

El genoma és un codi format per les seqüències de quatre nucleòtids representats per les lletres G, A, C i T. Des que es va conèixer, molta gent ha estat investigant com descodificar el genoma. I, per descomptat, per a descodificar, primer cal llegir aquestes lletres o, dit d'una altra manera, seqüenciar el genoma. És a dir, cal saber en quina ordre estan aquestes G, A, C, i aquestes T.
Per a això s'ha utilitzat sobretot un mètode en els últims 30 anys, si bé durant tots aquests anys ha experimentat millores significatives. Aquest mètode va ser desenvolupat per Frederick Sanger i els seus companys en la dècada dels 70 --pel que va rebre el segon Premi Nobel -.
La reacció bàsica del mètode de Sanger requereix quatre components principals: la porció d'ADN que es desitja seqüenciar o "patró d'ADN", quatre tipus de nucleòtids lliures, els petits filaments d'ADN de 20-30 nucleòtids coneguts com a iniciadors o primers, i l'enzim d'ADN polimerasa que sintetitza l'ADN.
La reacció comença escalfant el patró d'ADN perquè se separin els dos filaments. D'aquesta forma, l'iniciador s'unirà a un dels filaments (on hi ha una seqüència addicional). Perquè la polimerasa DNA allarga els filaments d'ADN, però no es pot començar pel no-res, per això és necessari l'iniciador. Una vegada que l'iniciador s'ha unit al patró d'ADN, l'ADN polimerasa iniciadora comença a estirar-se nucleòtid: on hi ha una A en el patró posarà una T i viceversa, i on hi ha una G una C i viceversa. Això ocorre, a més, sempre en un sentit determinat, és a dir, els dos extrems d'un filament d'ADN es denominen 5' i 3', i la polimerasa DNA afegeix nucleòtids a l'extrem 3'.
Per això, si partíssim de diverses còpies d'un patró d'ADN, obtindríem d'altres còpies d'un dels seus filaments. No obstant això, la clau del mètode de Sanger està en l'ús d'alguns nucleòtids modificats: els dideoxinucleótidos. A aquests nucleòtids transformats els falta un grup d'hidroxils en l'extrem 3', la qual cosa fa que no puguin afegir-se altres nucleòtids.

Quatre nucleòtids, quatre reaccions
El mètode de Sanger requereix quatre reaccions per a cada patró d'ADN. En les quatre reaccions es troben els quatre components esmentats anteriorment, però en cadascuna d'elles es col·loca un únic dideoxinucleótido. Així, per exemple, una de les reaccions contindrà quatre nucleòtids normals i alguns dideoxi-G. En aquesta reacció, quan l'ADN polimerasa iniciador comença a estirar-se, cada vegada que necessita un G, poden ocórrer dues coses aleatòries: Prendre una G normal o una dideoxi-G. La majoria són normals G, però quan presa un dideoxi-G, la polimerasa no podrà continuar estirant la cadena. Així, a partir de diverses còpies del patró, obtindrem filaments de diferent longitud, acabats tots en un G. I a partir de milions de còpies, finalment tindrem els filaments corresponents a cada G de la seqüència. I el mateix ocorre amb les altres tres reaccions.
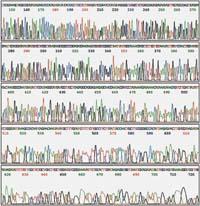
Posteriorment, els nous filaments obtinguts se separen per grandàries mitjançant una electroforesi. En l'electroforesi, els filaments d'ADN es col·loquen en un camp elèctric i com l'ADN té càrrega negativa es desplacen del pol negatiu al positiu. En el camí es col·loca alguna cosa que dificulti aquest moviment, com un gel porós, de manera que les parts grans triguen més temps a moure's d'un pol a un altre que les petites. D'aquesta forma, es poden distingir els filaments que tenen un costat d'un nucleòtid.
Per a poder visualitzar el resultat de l'electroforesi, els nous filaments han d'estar d'alguna manera marcats. Inicialment s'utilitzaven nucleòtids o iniciadors marcats radioactivament o fluorescents. Així, mitjançant una autoradiografía, o mitjançant raigs ultraviolats, es poden veure com a bandes, filaments separats per grandàries.
Cada reacció consisteix a traçar una línia en l'electroforesi, i el resultat final és que veurem una banda per cada nucleòtid de la seqüència en una de les quatre línies, i així, seguint l'ordre d'aquestes bandes, podrem llegir aquesta seqüència.

Automatització
Aquesta és bàsicament la tècnica utilitzada per a seqüenciar la major part de l'ADN seqüenciat fins al moment. Però des de la seva fundació, Sanger ha experimentat importants millores. Un dels més importants va ser la secuenciadora automàtica d'ADN inventada per Leroy Hood en 1986. Hood va marcar cadascun dels quatre dideoxinucleótidos de manera que proporcionés una fluorescència de diferent longitud d'ona (color). Així, d'una banda, en lloc de quatre reaccions, n'hi ha prou amb una sola, i per un altre, la longitud d'ona emesa en ser il·luminada amb raigs ultraviolats es pot detectar automàticament.
En els seqüenciadors automàtics, cada mostra va en una sola línia, detectant-se els quatre nucleòtids pel seu diferent color. En els seqüenciadors moderns, els filaments d'ADN se separen passant per uns capil·lars de fibra de vidre, a la sortida de l'extrem són colpejats per un làser i la màquina detecta la presència de fluoerescencia. Els filaments que salin primer seran l'iniciador més un nucleòtid i a continuació sortirà tota la seqüència.
Durant l'última dècada, els seqüenciadors automàtics d'ADN han millorat considerablement, podent processar moltes més mostres amb major rapidesa i facilitant el seu ús. En l'actualitat es poden seqüenciar centenars de mostres en una sessió i en un dia es poden realitzar unes 24 sessions.
Genoma gran

No obstant això, encara no és tasca fàcil descodificar tot un genoma. Les tècniques de seqüenciació de l'ADN serveixen per a seqüenciar petits fragments d'ADN, seqüències de 300-900 nucleòtids. De fet, en filaments més llargs, no poden separar-se amb precisió de la cara d'un nucleòtid.
Però el genoma petit d'un bacteri també conté milions de nucleòtids, i l'humà 3.000.000.000, dividit en 23 cromosomes. Imagina't que per a escriure tot el genoma lletra a lletra en aquesta revista necessitaríem unes 20.000 revistes. La longitud mitjana de les seqüències llegides (500 nucleòtids) seria el següent paràgraf, i en 20.000 revistes s'inclouen sis milions de paràgrafs d'aquest tipus. A més, per a aconseguir una fiabilitat suficient, una vegada no, caldria llegir entre 6 i 10 vegades totes aquestes revistes.
És el que van fer en el Projecte de Genoma Humà. Per parts van llegir tot el genoma humà lletra a lletra. No obstant això, dividir tot el genoma i llegir totes aquestes parts és una cosa i l'altra és saber l'ordre en el qual s'han d'unir aquestes parts.
El projecte va començar amb la construcció d'un mapa del genoma. Per a poder recórrer els cromosomes sense perdre, es van prendre milers de punts de referència. Una vegada realitzats aquests mapes, es va crear una "biblioteca" de fragments d'ADN que englobaria tot el genoma. Per a això, aquests trossos d'ADN van ser emmagatzemats a l'interior dels bacteris, i gràcies als punts de referència, cada part del genoma era coneguda. En definitiva, és el que ofereix qualsevol biblioteca: informació ordenada. Això ha permès treballar de manera coordinada en laboratoris de tot el món.
Els fragments d'ADN van ser emmagatzemats en els bacteris E. coli que viuen habitualment en els nostres intestins, com a cromosomes bacterians artificials (BAC). En cada BAC s'emmagatzemen fragments d'ADN de 100.000 o 200.000 nucleòtids.
Els bacteris E. coli conserven el temps que vulguis en el congelador. Així, quan el científic necessita un BAC de la biblioteca, el bacteri només ha de ressuscitar a 37 °C. A més, quan el bacteri que conté el fragment d'ADN es reprodueix amb ell. Així, n'hi ha prou que els bacteris deixin créixer una nit per a obtenir milions de còpies de la seva part interna d'ADN. A això se'n diu amplificació de l'ADN.
Els BACs són encara molt grans per a poder seqüenciar. Per això, els BACs també es divideixen aleatòriament, obtenint parts més petites que se solapen. Aquestes parts s'introdueixen en els virus o plasmidis que infecten els bacteris per a la seva posterior amplificació en els bacteris E. coli . Finalment, l'ADN dels bacteris és purificat i seqüenciat. A continuació s'identifiquen les seqüències que se solapen en les diferents parts i es completa la seqüència completa del BAC. Al mateix temps, els BACs també se superposen. I així, la lectura per lectura, el BAC per BAC i el cromosoma per cromosoma, fins a llegir tot el genoma.
Carrera genòmica
Llegir així un genoma complet requereix molt temps, treball i diners. Per això, els investigadors estan investigant contínuament per a desenvolupar tècniques de seqüenciació més ràpides i barates. Molts nous mètodes busquen augmentar la capacitat de seqüenciació i realitzar milers o milions de seqüències simultàniament. Però tampoc falten idees noves, com un mètode que detecta en el moment en què la dna-polimerasa afegeix el nucleòtid al filament, o que detecta corrents elèctrics que identificarien cada nucleòtid en passar els filaments per nanopors.
Els projectes són molts i s'estan desenvolupant a una velocitat vertiginosa. No és d'estranyar que les institucions públiques i privades estiguin invertint molt. En 2006, el National Human Genome Research Institute (NHGRI) dels EUA va aportar 13 milions de dòlars al finançament de projectes per a accelerar el desenvolupament de tecnologies que abarateixin la seqüenciació d'ADN. Aquest mateix any, la Fundació X Prize va anunciar un premi de deu milions de dòlars per al primer equip que en 2006 realitza un dispositiu capaç de seqüenciar cent genomes humans en deu dies. I, cent no, un projecte internacional que acaba de llançar pretén seqüenciar els genomes de mil éssers humans.
S'ha dit que la seqüenciació del genoma és un dels avanços més importants en la història de l'ésser humà. Investigadors de tot el món intenten revelar tots els secrets del genoma i descodificar el codi. És clar que estem en l'era de la genòmica.







