Genoma deskodetzen

Genoma kode bat da; G, A, C eta T letren bidez adierazten diren lau nukleotidoren sekuentziez osatutako kodea. Hori jakin zenetik, genoma nola deskodetu ikertzen aritu da jende asko. Eta, jakina, deskodetzeko, lehenengo irakurri egin behar dira letra horiek, edo, beste modu batera esanda, genoma sekuentziatu egin behar da. Hau da, G, A, C, eta T horiek zein ordenatan dauden jakin behar da.
Hori egiteko, azken 30 urteetan metodo bat erabili da batez ere, nahiz eta urte horietan guztietan hobekuntza nabariak izan dituen. Metodo hori Frederick Sanger-ek eta haren kideek garatu zuten 70eko hamarkadan --bigarren Nobel saria jaso zuen horregatik Sanger-ek--.
Sanger-en metodoaren oinarrizko erreakziorako, lau osagai nagusi behar dira: sekuentziatu nahi den DNA-zatia edo 'DNA patroia', lau motatako nukleotido askeak, 'abiarazle' edo primer izenez ezagutzen diren 20-30 nukleotidoko DNA-harizpi txikiak eta DNA sintetizatzen duen DNA polimerasa entzima.
Erreakzioa hasteko, DNA-patroia berotu egin behar da, bi harizpiak bereiz daitezen. Hala, abiarazlea harizpietako bati lotuko zaio (sekuentzia osagarria dagoen lekuan). Izan ere, DNA polimerasak luzatu egiten ditu DNA-harizpiak, baina ezin da ezerezetik hasi; horregatik behar da abiarazlea. Behin abiarazlea DNA patroiari lotu zaionean, DNA polimerasa abiarazlea luzatzen hasten da, nukleotidoz nukleotido: patroian A bat dagoen tokian T bat jarriko du, eta alderantziz, eta G bat dagoen tokian C, eta alderantziz. Hori, gainera, zentzu jakin batean gertatzen da beti; hau da, DNA-harizpi baten bi muturrei 5' eta 3' izena ematen zaie, eta DNA polimerasak 3' muturrari eransten dizkio nukleotidoak.
Horregatik, DNA patroi baten hainbat kopiatatik abiatuko bagina, patroi horren harizpietako baten beste hainbeste kopia lortuko genituzke. Baina, Sanger-en metodoaren gakoa nukleotido eraldatu batzuk erabiltzean dago: dideoxinukleotidoak. Nukleotido eraldatu horiei 3' muturrean hidroxilo-talde bat falta zaie, eta horren ondorioa da ezin zaiela beste nukleotidorik erantsi.

Lau nukleotido, lau erreakzio
Sanger-en metodoan, lau erreakzio egin behar dira DNA-patroi bakoitzerako. Lau erreakzioetan, goian aipatutako lau osagaiak daude, baina, bakoitzean, lau dideoxinukleotidotariko bakarra jartzen da. Hala, erreakzioetariko batek, esaterako, lau nukleotido normal eta dideoxi-G batzuk izango ditu. Erreakzio horretan, DNA polimerasa abiarazlea luzatzen hasten denean, G bat behar duen bakoitzean, bi gauza gerta daitezke zoriz: G normala edo dideoxi-G bat hartzea. Gehienak G normalak dira, baina, dideoxi-G bat hartzen duenean, polimerasak ezingo du katea luzatzen jarraitu. Hala, patroiaren hainbat kopiatatik abiatuta, luzera desberdineko harizpiak lortuko ditugu, guztiak G batean bukatuak. Eta milioika kopiatatik abiatuta, azkenean, sekuentziako G bakoitzari dagozkion harizpiak izango ditugu. Eta gauza bera gertatzen da gainerako hiru erreakzioetan ere.
Gero, lortutako harizpi berriak tamainaren arabera bereizten dira, elektroforesi baten bidez. Elektroforesian, eremu elektriko batean jartzen dira DNA-harizpiak, eta, DNAk karga negatiboa duenez, polo negatibotik positibora mugitzen dira. Bidean, mugimendu hori oztopatuko duen zerbait jartzen da --esaterako, gel porotsu bat--, eta, hala, zati handiek txikiek baino denbora gehiago behar izaten dute polo batetik bestera mugitzeko. Modu horretan, nukleotido bakarreko aldea duten harizpiak bereiz daitezke.
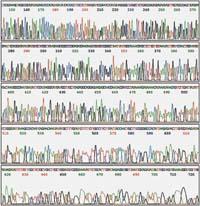
Elektroforesiaren emaitza ikusi ahal izateko, harizpi berriek nolabait markatuta egon behar dute. Hasieran, erradiaktiboki edo fluoreszenteki markatutako nukleotidoak edo abiarazleak erabiltzen ziren horretarako. Hala, autoerradiografia baten bidez, edo izpi ultramoreen bidez, banda gisa ikus daitezke, tamainaren arabera bereizitako harizpiak.
Erreakzio bakoitzarekin lerro bat egiten da elektroforesian, eta azken emaitza da sekuentziaren nukleotido bakoitzeko banda bat ikusiko dugula lau lerroetakoren batean; eta, hala, banda horien ordenari jarraituz, sekuentzia hori irakurri ahal izango dugu.

Automatizazioa
Hori da, funtsean, orain arte sekuentziatutako DNA gehiena sekuentziatzeko erabili den teknika. Baina, Sanger-ek sortu zuenetik, hobekuntza handiak izan ditu. Garrantzitsuenetako bat Leroy Hoodek 1986an asmatutako DNA automatikoki sekuentziatzeko makina izan zen. Hood-ek lau dideoxinukleotidoetariko bakoitza uhin-luzera (kolore) desberdineko fluoreszentzia emateko moduan markatu zuen. Hala, batetik, lau erreakzio egin beharrean, bakarra egitea nahikoa da, eta, bestetik, izpi ultramoreekin argitutakoan igortzen duten uhin-luzera automatikoki detekta daiteke.
Sekuentziadore automatikoetan, lagin bakoitza lerro bakarrean doa, eta kolore desberdinak dituztelako detektatzen dira lau nukleotidoak. Sekuentziadore modernoetan, DNA-harizpiak beira-zuntzezko kapilar batzuetatik pasaz bereizten dira, muturretik ateratzean laser batek jotzen ditu, eta makinak fluoereszentzia detektatzen du. Lehenengo aterako diren harizpiek abiarazlea gehi nukleotido bat izango dute, eta jarraian aterako da sekuentzia osoa.
Azken hamarkadan, DNA-sekuentziadore automatikoak asko hobetu dira: askoz lagin gehiago prozesa ditzakete, azkarrago, eta erabiltzeko errazagoak dira. Egun, ehunka lagin sekuentziatu daitezke saio batean, eta egun batean 24 bat saio egin daitezke.
Genoma handia

Hala ere, oraindik ez da lan makala genoma oso bat deskodetzea. DNAren sekuentziazio-teknikek DNA-zati txikiak sekuentziatzeko balio dute, 300-900 nukleotidoko sekuentziak. Izan ere, hori baino harizpi luzeagoetan, ezin dira zehaztasunez bereizi nukleotido bakarreko aldearekin.
Baina bakterio baten genoma txikiak ere milioika nukleotido ditu; eta gizakiarenak 3.000.000.000, 23 kromosomatan banatuta. Pentsa, aldizkari honetan letraz letra genoma osoa idazteko, 20.000 bat aldizkari beharko genituzke. Irakurritako sekuentzien batez besteko luzera (500 nukleotido) paragrafo hau litzateke gutxi gorabehera; eta 20.000 aldizkaritan honelako sei milioi paragrafo sartzen dira. Gainera, fidagarritasun nahikoa lortzeko, behin ez, 6-10 aldiz irakurri beharko lirateke aldizkari horiek guztiak.
Horixe egin zuten, hain zuzen ere, Giza Genoma Proiektuan. Zatika-zatika giza genoma osoa irakurri zuten letraz letra. Baina, genoma osoa zatitu eta zati horiek guztiak irakurtzea gauza bat da, eta bestea, gero zati horiek zein ordenatan elkartu behar diren jakitea.
Proiektua hasteko, genomaren mapa bat eraiki zuten lehenengo. Kromosometan zehar galdu gabe ibili ahal izateko, milaka erreferentzia-puntu hartu zituzten. Behin mapa horiek eginda, genoma osoa bilduko zuen DNA-zatien 'liburutegi' bat sortu zuten. Horretarako, DNA-zati horiek bakterioen barnean gorde zituzten, eta erreferentzia-puntuei esker jakin zezaketen zati bakoitza genomaren zein tokitakoa zen. Azken finean, hori da edozein liburutegik eskaintzen duena: informazio ordenatua. Horri esker, mundu osoko laborategietan modu koordinatuan lan egin ahal izan dute.
DNA-zatiak gure hesteetan bizi ohi diren E. coli bakterioetan gorde zituzten, bakterio-kromosoma artifizial (BAC) gisa. BAC bakoitzean, 100.000 edo 200.000 nukleotidoko DNA-zatiak gordetzen dira.
E. coli bakterioak nahi adina denbora gorde daitezke izozkailuan. Hala, zientzialariak liburutegiko BAC bat behar duenean, bakterioak 37 ºC-an berpiztu besterik ez du egin behar. Gainera, DNA-zatia barruan duen bakterioa ugaltzen denean, DNA-zati hori ere harekin batera ugaltzen da. Hala, bakterioei gau batez hazten uztea nahikoa da barruan duten DNA-zatiaren milioika kopia lortzeko. Horri DNA anplifikatzea esaten zaio.
BACak oso handiak dira, oraindik, sekuentziatu ahal izateko. Horregatik, BACak ere zatitu egiten dira zoriz, eta gainjartzen diren zati txikiagoak lortzen dira. Zati horiek bakterioak infektatzen dituzten birusetan edo plasmidoetan sartzen dira, gero, berriz ere, E. coli bakterioetan anplifikatzeko. Azkenik, bakterioetatik DNA purifikatu eta sekuentziatu egiten da. Gero, zati desberdinetan gainjartzen diren sekuentziak identifikatu eta BACaren sekuentzia osoa osatzen da. Aldi berean, BACak ere gainjarri egiten dira bata bestearekin. Eta, hala, irakurketaz irakurketa, BACez BAC eta kromosomaz kromosoma, genoma osoa irakurri arte.
Lasterketa genomikoa
Genoma oso bat horrela irakurtzeko, denbora, lan eta diru asko behar da. Horregatik, ikertzaileak etengabe ari dira sekuentziazio-teknika azkarragoak eta merkeagoak garatzeko ikertzen. Metodo berri askok sekuentziazio-ahalmena handitzea bilatzen dute, eta milaka edo milioika sekuentziazio aldi berean egitea. Baina ideia berriak ere ez dira falta; esaterako, DNA-polimerasak nukleotidoa harizpiari eransten dion unean bertan detektatzen duen metodo bat, edo harizpiak nanoporoetatik pasatzean nukleotido bakoitza identifikatuko luketen korronte elektrikoak detektatzen dituena.
Proiektuak asko dira, eta zorabiatzeko moduko abiaduran ari dira garatzen. Ez da harritzekoa, erakunde publikoak eta pribatuak inbertsio handiak egiten ari dira. 2006an, AEBko National Human Genome Research Institute-k (NHGRI) 13 milioi dolar jarri zituen hainbat proiektu finantzatzeko, DNA sekuentziazioa merkeagoa egingo duten teknologien garapena azkartzeko asmoz. Urte berean, X Prize fundazioak hamar milioi dolarreko saria iragarri zuen, 2006an ehun giza genoma hamar egunetan sekuentziatzeko gai den gailua egiten duen lehen taldearentzat. Eta, ehun ez, mila gizakiren genomak sekuentziatzea du helburu berriki abiarazi duten nazioarteko proiektu batek.
Esan izan da genomaren sekuentziazioa gizakiaren historian egin den aurrerapausorik garrantzitsuenetako bat dela. Mundu osoko ikertzaileak buru-belarri ari dira genomaren sekretu guztiak argitu nahian, kodea deskodetu nahian. Argi dago, genomikaren aroan gaude.






