Europako ezti-erleen diagnostiko genetikorako tresnak
Apis mellifera edo ezti-erlea munduko polinizatzaile garrantzitsuena da gaur egun, bai ekologikoki bai ekonomikoki, eta maiz erabiltzen da eztia ekoizteko eta nekazaritza-laboreak polinizatzeko. Hala ere, erle hauek mehatxupean daude hainbat faktoreren ondorioz. Haien artean, dibertsitate genetikoaren murrizketak eta ingurunera egokitutako tokiko subespezieen galerak eragin nabarmena dute, bai eskala handiko erreginen hazkuntza eta inportazio komertzialak bai distantzia luzeko erlezaintza migratzaileak bultzatuta. Munduko erle-populazioaren osasuna bermatzeko, ezinbestekoa da tokiko subespezieen kontserbazioa ziurtatzea, bai eta haien herentzia naturala eta egokitzeko gaitasuna mantentzea ere. Europa ezti-erleen ondare genetikoan aberatsa da (espeziean definitutako 5 leinu ebolutiboetatik gutxienez 4 leinutako 13 subespezieren ordezkariak ditu), eta funtsezko eskualdea suertatu da kontserbaziorako eta berau erraztuko duten tresnak garatzeko. Hori dela eta, Europako SmartBees proiektuaren esparruan, ezti-erle europarren subespezieen identifikazioa ahalbidetuko duen DNA mikrotxipa garatu da. Diagnostiko-tresna horri esker kontserbazio jasangarria, erreginen merkataritza kontrolatua, erlezainen produktuen egiaztapena eta tokiko erleen ondare genetikoa zaintzen duten hazkuntza-jarduerak erraztuko eta bultzatuko dira.
Ezti-erleen aniztasuna babestea beharrezkoa da
Ezti-erleak (Apis melllifera) ekosistema anitzetan daude, zenbait eboluzio-leinutan eta gutxienez 30 subespezietan banatuta. Europan aniztasun horren zati handi bat dago, eta subespezie endemiko ugari ditu lau leinutan banatuta: Afrikakoa (A), Europako erdialdekoa eta ekialdekoa (C), Europako mendebaldeko eta iparraldekoa (M) eta Ekialde Hurbileko eta Asiako erdialdekoa (O). Hala ere, giza jarduerak, pixkanaka, murriztu egin ditu bai Europako ezti-erleen dibertsitate genetikoa eta bai haien lurralde naturala. Hain zuzen ere, erreginen merkataritza eta inportazioa eta distantzia luzeko transhumantzia direla medio, dagoen beldurretako bat zera da, ingurunera egokitutako ezti-erle autoktonoen populazioak murriztu edo galdu egingo direla, autoktonoak ez diren erleek ordezkatuta edo hibridazio hutsagatik. Frogatuta dago ingurunera egokituta dauden ezti-erleek irauteko gaitasun handiagoa dutela. Beraz, garrantzitsua da erleen hazkuntza sustatzea erleak epe luzera iraungo badu.
Europan ezti-erle autoktonoak kontserbatzeko eta hazteko proiektu ugari jarri dira abian; haien artean, hobekuntza genetikorako programak. Arrakastatsuak izango badira, ernalketa-esparrua kontrolatu beharra dago, eta populazioaren jatorri genetikoaren jarraipena egin. Alde horretatik, beharrezkoa da analisi genetiko azkar, zehatz eta erabilerraza egiteko tresna bat.
SmartBees proiektua helburu batekin abiatu zen: tresna molekular berriak garatzea, aukera emango zutenak Europako ezti-erleen aniztasuna deskribatzeko eta kontserbatzeko. Hala, erlearen laginketa sakon bat eginda, tresna bat diseinatu dugu, nukleotido bakarreko markatzaile genomikoz osatua; horri esker, haren dibertsitate genetikoa deskriba daiteke (SNP), eta Europako edozein erleren subespeziea identifikatu.
Prozedura
22 populazio lagindu dira (1. irudia), Europako lau leinu ebolutiboen eta Europan eta inguruko eskualdeetan aurki daitezkeen 14 subespezieen ordezkari direnak. Populazioak lagintzeko, elkarrekin erlaziorik ez duten erlauntzetako 100 erle langile hartu dituzte. Guztira, 2.000 lagin baino gehiago, orain arte Europako ezti-erlearekin egindako laginketarik osoena.
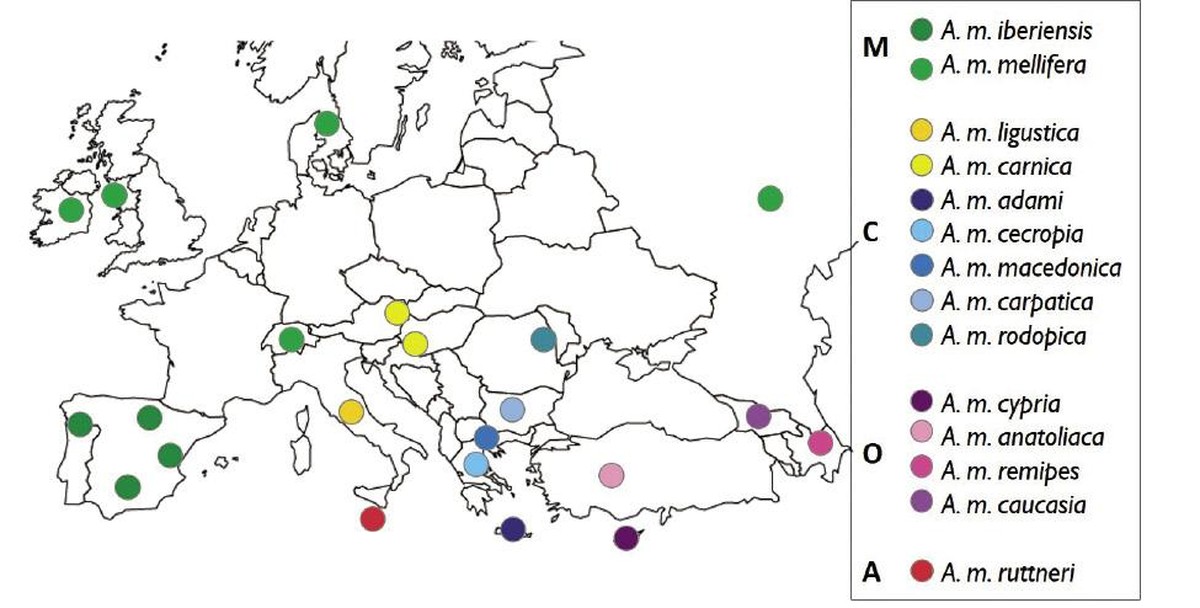
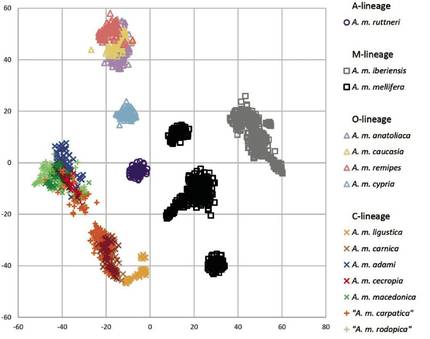
Laginak jatorrizko populazioaren arabera multzokatu ostean, haien DNA erauzi eta sekuentziatu da. Sekuentziazioaren datuetan oinarrituta, jatorriari buruz informazio gehien ematen duten markatzaile genetikoak hautatu dira. Hau da, Europako ezti-erleen 14 subespezieak hobekien bereizten dituzten markatzaile genetikoak; guztira, nukleotido bakarreko 4.000 polimorfismo (SNP) baino gehiago. Genotipazioaren emaitzak t-SNE izeneko diagrama erabiliz irudikatu dira. Diagrama horrek laginak ahalik eta modurik trinkoenean multzokatzen ditu bi dimentsioko mapa batean, banako bakoitza ikur batez ordezkatuz (2. irudia). Metodo hori erabiliz, beren eboluzio-leinuaren edo subespeziearen arabera multzokatu dira lagindutako erleak talde bakartuetan (puntu-hodeiak). Afrikako leinutik lagindutako subespezie bakarra, A. m. ruttneri, grafikoaren erdian kokatu da, gainerako multzoen artean. O leinuan, A. m. cypria erleak erabat bananduta agertu dira elkarrekiko hain ezberdinak ez diren hiru erlerengandik (A. m. anatoliaca, A. m. caucasia eta A. m. remipes). M leinuko bi subespezieak ondo bereizi dira, A. m. mellifera-ren populazioak hiru azpimultzotan bilduz. Azpimultzo horiek laginketa-eskualde urrunak irudikatzen dituzte (Burzyan eskualdea, Errusia, A. m. melliferaren kluster gorena 1. irudian), edo eskualde bakartuak (Læsla uhartea, Danimarka, A. m. melliferaren behe klusterra). C leinuko laginak hiru azpitaldetan banatu dira: (i) A. m. ligustica, (ii) A. m. carnica erleak zenbait A. m. carpatica erlerekin batera eta (iii) A. m. macedonica, A. m. cecropia, A. m. adami, A. m. rodopica eta gainerako A. m. carpatica erleez osatutako azpitalde heterogeneoa.
Saiakuntza gehigarriak eta ikaskuntza automatikoaren eredua
Horietaz gainera, Europa osoko beste 1.900 erle genotipatu dira, SmartBeesen hazkuntzarako erlategietatik lortuta. 1.900 lagin horiekin, lehendik genotipatutako 2.000 laginekin batera (guztira 3.900 lagin baino gehiago), eredu estatistiko bat eratu dute, Europako ezti-erleak sailkatzen dituena ikaskuntza automatikorako algoritmoak erabiliz. Eredu horri esker, kalkula daiteke lagin batek zer probabilitate duen Europako 14 subespezieetakoa izateko. Ikaskuntza-eredu automatikoek alde on bat dute, ez direla oinarritzen aldez aurreko hipotesietan, eta, ondorioz, desberdintasun sotilak detekta ditzakete. Ezaugarri hori bereziki garrantzitsua izan da gure azterketarako, modua eman baitigu lotura genetiko estua duten azpiespezie-kopuru handiok elkarrengandik bereizteko. Jatorrizko artikuluan (Momeni et al. 2021), sailkapen-metodo espezifikoei buruzko xehetasun gehiago aurki daitezke.
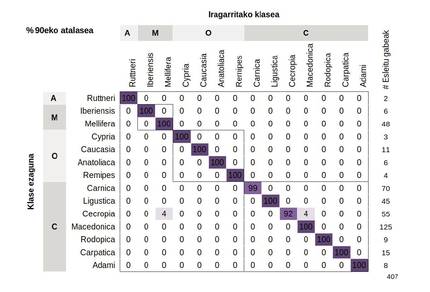
Zehaztasuna azpiespezieak esleitzean
Ereduak zuzen esleitu ditu lagin gehienak, % 96,2ko batez besteko zehaztasunarekin. Emaitza horiek bistaratzeko modurik onena nahasmen-matrize bat da; aldez aurretik zuzen edo oker sailkatutako laginen portzentajeak adierazten ditu eredu horrek (3. irudia). Diagonal zentralaren zifrek ondo sailkatutako laginen ehunekoa adierazten dute; goiko eta beheko triangeluek, berriz, oker sailkatutako laginak adierazten dituzte. Oker sailkatutakoa izango da ereduak iragarritako laginak eta etiketatutakoak azpiespezie desberdinak badira. Errore horiek agertu dira, batetik, saiakuntzetako laginen etiketatzeak okerrak izan direnean eta, bestetik, erreferentziazko populazioen arteko bereizketak oso txikiak izan direnean, dela hurbiltasun geografikoagatik, dela giza interferentziengatik.
Eredu hori kontserbazio- eta hazkuntza-programetan aplikatua izateko, % 90eko gutxieneko atalasea ezarri dugu. Gauzak horrela, lagin baten pronostikoa % 90eko atalasetik behera badago, “ez esleituta” bezala sailkatuko da. Muga gainditzen badu, berriz, dagokion subespezieari esleituko zaio.
Aniztasunaren erronkak eta hurrengo urratsak
Europako ezti-erleen aniztasuna erronka handia izan da subespezieen diagnostikorako tresna bat diseinatzeko orduan. Eboluzio-leinuen dibergentzia handiari esker, bereizketa genetikoa erraza izan da, SNP gutxi batzuk erabiliz. Alabaina, zailagoa izan da subespezieak bereiztea, eboluzio-leinu bereko subespezieen arteko dibergentzia duela gutxi gertatu delako eta, hortaz, genetikoki oso antzekoak direlako. Gainera, Europako zenbait eskualdetan, zehaztu gabe daude A. mellifera subespezieen aldakuntzaren gorabeherak, eta, kanpoko erleen, sarrera artifizialak lausotu egin ditu subespezieen arteko muga naturalak. Baliteke, halaber, hazkuntza-programa nazionalek geneen fluxu naturala oztopatu izana eta jatorrizko subespeziearen ondare genetikoa aldatu izana. Horren ondorioz, subespezie batzuk erraz sailkatu dira gure diagnostiko-tresna erabiliz, eta beste lagin batzuk, berriz, esleitu gabe geratu dira. Hala ere, tresna dinamiko moldagarria da, eta aukera ematen du erreferentziazko datu-basea hobetzeko eta/edo azpiespezie gehiago sartzeko. Ildo horretan, abian dauden ikerketak kontuan izanda, metodoa Siziliako A. m. sicilianari aplika dakioke.
Erabilgarritasuna
Tresna honekin, erle-lagin gehiagoren jatorri genetikoa zehaztu ahalko da, eta hori ezinbestekoa suertatuko da arlo ugaritan: erlezainek beren erleen subespeziea eta hibridazio-maila zehaztu ahalko dute, eta beren produktuei abala eman; Europako kontserbazioaren arduradunek gordailuetan dauden erlauntzen hibridazio-tasak kontrolatu ahalko dituzte; albaitariek erreginen merkataritza kontrolatu ahalko dute; eta erleen hazleek beren erreginen subespeziea ziurtatu.
Argitalpen zientifiko osoa
Eskerrak
Erlezainen, erle-hazleen eta laguntzaile ugariren laginekin gauzatu dugu ikerketa hau. Gure eskerrik beroenak ematen dizkiegu beren laguntzagatik. SmartBees proiektua Europako Batzordeak finantzatu du, FP7 KBBE programaren barruan (2013.1.3-02, Grant nr. 613960). Melanie Parejok Eusko Jaurlaritzaren dirulaguntza jaso du (IT1233-19).
Idatzi zuk zeuk Gai librean atalean
Gai librean aritzeko, bidali zure artikulua aldizkaria@elhuyar.eus helbidera
Hauek dira Gai librean atalean Idazteko arauak
Gai honi buruzko eduki gehiago
Elhuyarrek garatutako teknologia










